
Um estudo sobre o câncer de bexiga conduzido pelo Laboratório de Bioinformática da UFPR foi selecionado pela revista Cell para publicação em sua edição de setembro deste ano. O periódico de alcance internacional é um dos mais destacados no campo da biologia molecular e tem sido protagonista na divulgação de descobertas na área da oncologia.
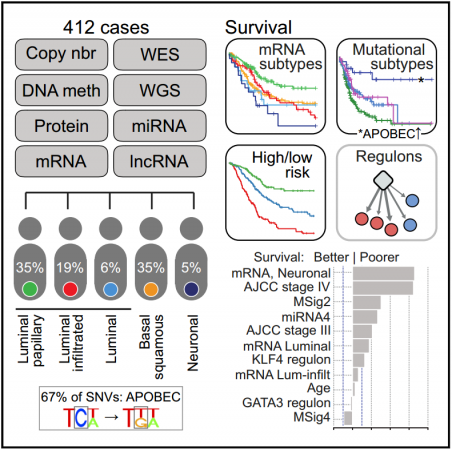
O estudo revelou que o câncer de bexiga se divide em 5 subtipos com formas específicas de desenvolvimento. Segundo os pesquisadores, a descoberta pode ter um grande impacto para o desenvolvimento de novos protocolos clínicos e diretrizes terapêuticas, pois cada sub-categoria apresenta um modo diferente de resposta aos tratamentos da doença. As informações podem ser utilizadas também para o desenvolvimento de novas drogas.
A equipe do laboratório da UFPR foi liderada pelo professor Mauro Antônio Alves Castro, que analisou um vasto conjunto de dados ligados a 412 pacientes com este tipo de câncer, que mata todos os anos cerca de 150 mil pessoas. O banco de dados foi gerado pelo consórcio internacional “The Cancer Genome Atlas” (TCGA) que até este ano recolheu informações de cerca de 11 mil casos de câncer na busca por gerar um banco de dados com informações variadas que podem ser base de uma diversidade de estudos. Até agora cerca de mil trabalhos sobre o tema utilizaram estas informações. Em 2015 a equipe da UFPR passou a integrar oficialmente o grupo.


A quantidade de dados gerados, mais de dois petabytes até agora, é muito maior do que a capacidade de análise das instituições que lideram o consórcio e é justamente onde entra a importância do trabalho feito na UFPR. “Nosso grupo na UFPR desenvolveu métodos computacionais que despertaram o interesse dos líderes deste consórcio, que nos convidaram a integrar os esforços na caracterização da coorte de câncer bexiga. Nossa participação foi estratégica, pois contribuímos com métodos para interpretar a grande quantidade de dados gerados pelo consórcio” explica o pesquisador. Uma “coorte”, em uma explicação aproximada, é o conjunto de pacientes com certo tipo de doença que são investigados ao longo do tempo.
A pesquisa no Brasil, que além da equipe da UFPR, teve a participação do estatístico Benilton de Sá Carvalho, professor da Universidade Estadual de Campinas (Unicamp), foi responsável principalmente por identificar quais genes tem um maior impacto no desenvolvimento do câncer de bexiga. A chamada “caracterização molecular de reguladores transcricionais” reúne diversas informações relacionadas a estes genes e a atuação das proteínas que eles produzem. Informações como estas são essenciais para o desenvolvimento de novas drogas utilizadas na terapia.
Outra importante contribuição foi a análise dos dados de sobrevida dos pacientes da coorte. Estes dados ajudam a compreender como será o desenvolvimento do tumor, o que é essencial para o tratamento.


O estudo também ajudou a descrever cinco subtipos deste câncer, cujas características estão descritas no artigo. Com todas estas informações o laboratório pode estabelecer os fluxos de análise para a caracterização de cada tumor, uma espécie de roteiro que os médicos podem seguir para identificar com mais exatidão qual tipo de tumor está se desenvolvendo e buscar um tratamento mais adequado.
Além de Castro e Cardoso, o pesquisador Vinicius de Saraiva Chagas, então estudante do Programa de Pós-graduação em Bioinformática e hoje Cientista de Dados no Hospital Albert Einstein, também é coautor do estudo. Foi dele a primeira proposta de trabalhar com o tema. Também compõe a equipe do laboratório a engenheira de bioprocessos e biotecnologia Clarice dos Santos Groeneveld, a biomédica Kelin Gonçalves de Oliveira e a bioinformata Sheyla Trefflich.
TCGA
Foi em 2015 que o trabalho da equipe da UFPR chamou a atenção do consórcio internacional, TCGA. Um método desenvolvido pelos pesquisadores do Laboratório de Bioinformática traçou variações genéticas do câncer de mama, o que resultou em uma publicação em outro conceituado periódico internacional naquele ano, a Nature Genetics. O estudo utilizou informações do TCGA, que são abertas para acesso de pesquisadores. A qualidade dos métodos desenvolvidos no laboratório resultou no convite para que a UFPR passasse a integrar o grupo de análise dos dados de câncer de bexiga.
Em 2017 o consórcio encerrou as atividades ligadas a este banco de dados, mas a parceria que envolve diversas instituições e é liderada pelo Instituto Nacional de Saúde (National Institute of Health) dos Estados Unidos, já articula um novo consórcio que deve trabalhar com um volume de dados e número de pacientes ainda maiores. O laboratório da UFPR também se prepara para acompanhar desde o início a implementação deste novo projeto.
A equipe de jornalismo da UFPR falou com o professor sobre o estudo, confira!
Aline Fernandes França – Como funciona o consórcio internacional “The Cancer Genome Atlas” e qual a participação da UFPR?


Mauro Antônio Alves Castro – O TCGA é uma iniciativa do Instituto Nacional do Câncer (NCI) e do Instituto Nacional de Pesquisa do Genoma Humano (NHGRI) dos Estados Unidos, que gerou mapas abrangentes e multidimensionais das principais alterações genômicas em 33 tipos de câncer, de mais de 11.000 pacientes. O conjunto de dados do TCGA, composto atualmente por mais de dois petabytes de dados genômicos, foi disponibilizado publicamente, e essa informação genômica tem ajudado a comunidade científica a melhorar a prevenção, o diagnóstico e o tratamento do câncer.
Estes dados já contribuíram com mais de mil estudos na área do câncer, conduzidos tanto por pesquisadores independentes como pela rede de pesquisa do TCGA. Nosso grupo na UFPR desenvolveu métodos computacionais que despertaram o interesse dos líderes deste consórcio, que nos convidaram a integrar os esforços na caracterização da coorte de câncer bexiga. Nossa participação foi estratégica, pois contribuímos com métodos para interpretar a grande quantidade de dados gerados pelo consórcio.
AFF – Qual o significado das descobertas publicadas na revista Cell?
MAAC – O câncer de bexiga causa cerca de 150 mil mortes por ano no mundo. Não é uma mas múltiplas doenças, e sabemos que existem variações que podem afetar a resposta ao tratamento e a sobrevivência a longo prazo. Este estudo fornece uma compreensão mais ampla e refinada dos fundamentos moleculares associados a essas variações. Em 2014, este mesmo grupo de pesquisadores publicou na revista Nature os resultados preliminares desta coorte de pacientes, na ocasião com 131 tumores de bexiga. O trabalho atual expandiu o estudo de 2014 com uma coorte três vezes maior, e refinou os subtipos moleculares com a integração de mais tipos de dados genômicos.
AFF – Qual o método utilizado no estudo da caracterização molecular do câncer de bexiga?
MAAC – Foram muitos. A principal característica deste tipo de estudo é a geração de dados multidimensionais, ou seja, diversos aspectos do tumor são investigados para o mesmo conjunto de amostras, tais como o genoma, o transcriptoma, o proteoma, padrão de mutação, dados clínicos e sobrevida.
AFF – Quem integrou o grupo de pesquisa?
MAAC – A equipe de Brasileiros foi composta por dois pesquisadores (um da UFPR e outro da Unicamp) e um aluno do mestrado do curso de Pós-graduação em Bioinformática da UFPR (Vinicius de Saraiva Chagas). Nós conduzimos a análise de sobrevida, bem como assinamos a caracterização das redes de regulação gênica. Atualmente nossa equipe conta com novos integrantes envolvidos na continuidade desta colaboração. Trabalhamos com um time de pesquisadores muito experiente, sendo nosso principal contato o Dr. Gordon Robertson, do Michael Smith Genome Sciences Centre (BC Cancer Agency, Vancouver, Canadá).
Rodrigo Choinski – A pesquisa já vinha sendo desenvolvida no Laboratório de Bioinformática da UFPR mesmo antes de vocês integrarem o estudo do câncer de bexiga. Além da questão dos métodos desenvolvidos no laboratório (apresentados previamente no estudo sobre câncer de mama), como aconteceu a entrada nesta pesquisa em especial?
MAAC – Em 2014 foi publicado na Nature um artigo importante deste grupo, que era uma prévia da coorte com 131 pacientes. Quando uma coorte é muito extensa, demora muito tempo para seguir os pacientes. Você tem um volume de dados que ainda não é o volume de dados final, aí você antecipa o estudo e apresenta os resultados estáveis, em 2014 eles fizeram essa publicação.
Naquela ocasião o grupo ainda não tinha os dados de sobrevida porque a coorte estava em andamento, estas coortes são longitudinais, você tem que acompanhar ao longo do tempo. Agora nós tivemos a oportunidade de conduzir a análise de sobrevida, em uma colaboração com um pesquisador da Unicamp, o estatístico Benilton de Sá Carvalho.
RC – Quais outras contribuições para o estudo?
MAAC – Outra contribuição foi a caracterização molecular de reguladores transcricionais, uma parte da pesquisa que interpreta os dados dos tumores do ponto de vista de mecanismos. Nem todos os nossos genes contribuem para uma determinada doença, na verdade é uma minoria deles. Existem genes que são mais importantes do que outros, que controlam eventos como proliferação e metástase. Um dos objetivos de nossa análise foi identificar genes que eram capazes de contribuir de maneira significativa para o desenvolvimento dos tumores, o que nós chamamos de genes regulatórios. Fizemos então a caracterização molecular deste genes.
RC – Qual o impacto disto para o desenvolvimento de novos tratamentos?
MAAC – Um dos aspectos mais promissores dessa abordagem é que quando você identifica genes regulatórios associados a um tipo de câncer, você cria a possibilidade de desenvolver drogas visando pontos importantes no controle do tumor . O primeiro passo para o desenvolvimento de uma droga é pensar onde estamos mirando, e essa abordagem consegue listar bons candidatos para isso.
RC – Um dos aspectos desenvolvidos pelo estudo, em destaque no artigo, é a subtipagem, o professor poderia explicar como estas descobertas podem ajudar no tratamento?
MAAC – Mesmo que uma pessoa seja diagnosticada com o mesmo tipo de doença, como o câncer de bexiga, por exemplo, ela se apresenta de formas distintas, você vai ter subtipos deste tipo de câncer. As informações que geramos podem ser utilizadas para caracterizar subtipos, assim é possível investigar se é um câncer mais agressivo, tentar antecipar qual será a resposta a uma determinada droga, entre outros desenvolvimentos. A subtipagem molecular proporciona um refinamento na classificação do tumor. Uma coisa é você ter boas drogas para combater o tumor, outra coisa é você identificar quem pode se beneficiar destas drogas.
AFF – Quais as perspectivas futuras para a pesquisa?
MAAC – Um conjunto de dados desta natureza é uma contribuição que deve dar origem a muitos outros estudos, pela extensão e complexidade das possíveis associações ainda não identificadas. Estamos atualmente dando seguimento aos métodos computacionais que desenvolvemos nesta parceria e esperamos transferir para outros tipos de neoplasias.
por Aline Fernandes França e Rodrigo Choinski






