Código genético pode ajudar no aumento da produção agrícola do Brasil
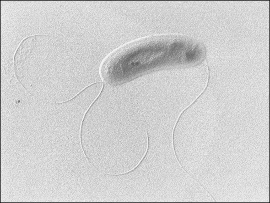
O volume e a qualidade da produção agrícola do Brasil podem aumentar com a aplicação da biotecnologia desenvolvida por pesquisadores que integram o Projeto Genoma do Paraná (GENOPAR). Desde 2002, cerca de 140 cientistas estiveram envolvidos no seqüenciamento de 5.079 genes da bactéria Herbaspirillum seropedicae. O microorganismo funciona como um adubo natural e pode gerar uma economia anual ao Brasil de R$ 850 milhões em fertilizantes.
“A Herbaspirillum seropedicae é capaz de produzir amônia a partir do nitrogênio da atmosfera”, explica o Dr. Fábio de Oliveira Pedrosa, professor da UFPR e coordenador do projeto. Segundo ele, a utilização dos recursos genéticos da bactéria pode estimular o crescimento da planta e a produtividade de grãos. “Quando o agricultor consegue uma melhoria de 5% na safra já comemora, imagine a repercussão quando conseguirmos aumentar a produtividade de 20 a 30%”, fala Pedrosa, animado com os benefícios sociais da descoberta.
A bactéria pode ser encontrada no interior de diversas espécies de interesse agrícola – como milho, trigo, sorgo, cana-de-açúcar e bananeira – e não é patogênica para o ser humano, plantas ou animais. Ela foi identificada na década de 1980, por pesquisadores da Embrapa-Agrobiologia (Empresa Brasileira de Pesquisa Agropecuária), e desde então tem despertado o interesse da comunidade científica, por seu grande potencial econômico e ambiental.
Tecnologia
Criado para seqüenciar o genoma da Herbaspirillum seropedicae, o GENOPAR foi estruturado como uma rede estadual de laboratórios composta por 12 unidades. Apesar do trabalho estar distribuído em todo o Paraná, a coordenação é realizada pelo Núcleo de Fixação de Nitrogênio do Departamento de Bioquímica e Biologia Molecular da UFPR. “Temos a segunda maior rede estadual de pesquisa genômica do Brasil e somos altamente qualificados, tanto em infra-estrutura como em recursos humanos”, afirma Pedrosa.
A identificação dos genes foi realizada utilizando a bioinformática, especialmente o software BLAST, que compara seqüências da H. seropedicae com seqüências de outros genomas presentes nos bancos de dados do Centro Nacional para Informação Biotecnológica dos Estados Unidos. “Utilizamos um programa de computador para remontar a estrutura do DNA, que é fragmentada durante o processo, porque a bactéria em estudo possui 5,5 milhões de bases”, explica Pedrosa. “A partir da ordenação dos fragmentos, reconstituímos o genoma original com tecnologia computacional e identificamos as funções dos genes encontrados por comparação com outros genomas.”
As funções de 57% dos genes da H. seropedica já foram identificadas pelo GENOPAR. Os outros 43% não têm função conhecida e constituem um tesouro biológico que ainda deve ser explorado. Segundo Pedrosa, a análise detalhadas dos 5 mil genes identificados revela genes e vias metabólicas que têm funções ou formam produtos de interesse farmacológico e industrial. No entanto, com as informações já disponíveis, o futuro parece promissor. “Com o que foi identificado da H. seropedicae é possível criar variedades de plantas mais resistentes à seca, biofertilizantes mais eficientes e não nocivos ao meio ambiente”, conclui Pedrosa.
Foto(s) relacionada(s):
Link(s) relacionado(s):
Fonte: Jackson Gomes Júnior (Funpar)